Badacze z UW we współpracy z japońskimi naukowcami prowadzą badania ryboprzełączników – segmentów RNA, które kontrolują produkcję białek w bakteriach. Artykuł na ten temat ukazał się w czasopiśmie „Proceedings of the National Academy of Sciences” (PNAS).
Naukowcy z UW – prof. Joanna Trylska, Piotr Chyży oraz Marta Kulik – we współpracy z grupą prof. Yujiego Sugity z RIKEN (Rikagaku Kenkyūjo), instytutu badawczego w Japonii, przeprowadzili symulacje dynamiki molekularnej dla układów neomycyna-ryboprzełącznik. Zostały one wykonane przy użyciu superkomputerów na Uniwersytecie Warszawskim (w Centrum Nowych Technologii oraz Interdyscyplinarnym Centrum Modelowania Matematycznego i Komputerowego) oraz w RIKEN Advanced Center for Computing and Communication.
Ryboprzełączniki
Ryboprzełączniki to fragmenty informacyjnego RNA (mRNA) znajdujące się tuż przed częścią mRNA kodującą białko. Wyłączają produkcję białka, gdy zwiąże się z nimi odpowiednia cząsteczka, zwykle metabolit. Wpływanie na stan ryboprzełączników daje kontrolę nad produkcją konkretnych białek niezbędnych do życia komórki.
– Naturalnie występujące ryboprzełączniki mogą być celami dla nowych antybiotyków. Jeśli udałoby się zaprojektować cząsteczki, które blokowałyby określony ryboprzełącznik w bakteriach chorobotwórczych, mogłoby to prowadzić do unicestwienia tych komórek. Ryboprzełączniki nie występują jednak we wszystkich komórkach. Dlatego, szczególnie w komórkach ludzkich, interesujące są syntetyczne ryboprzełączniki, które można wprowadzić do mRNA i w ten sposób uzyskać kontrolę nad produkcją określonych białek. Projektowanie syntetycznych ryboprzełączników jest jednak trudne, gdyż wymaga przewidzenia, jak zmieni się ich struktura po związaniu się z nową cząsteczką kontrolującą stan przełącznika. Sam fakt, że cząsteczka silnie wiąże się z ryboprzełącznikiem, nie oznacza, że zadziała on zgodnie z oczekiwaniami w komórkach – mówi prof. Joanna Trylska z Centrum Nowych Technologii UW.
Wiązanie cząsteczek
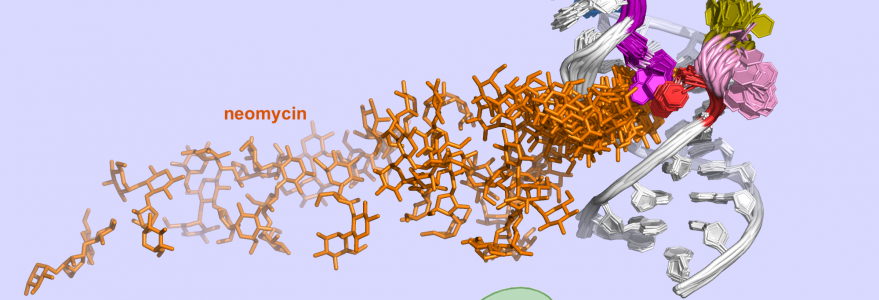
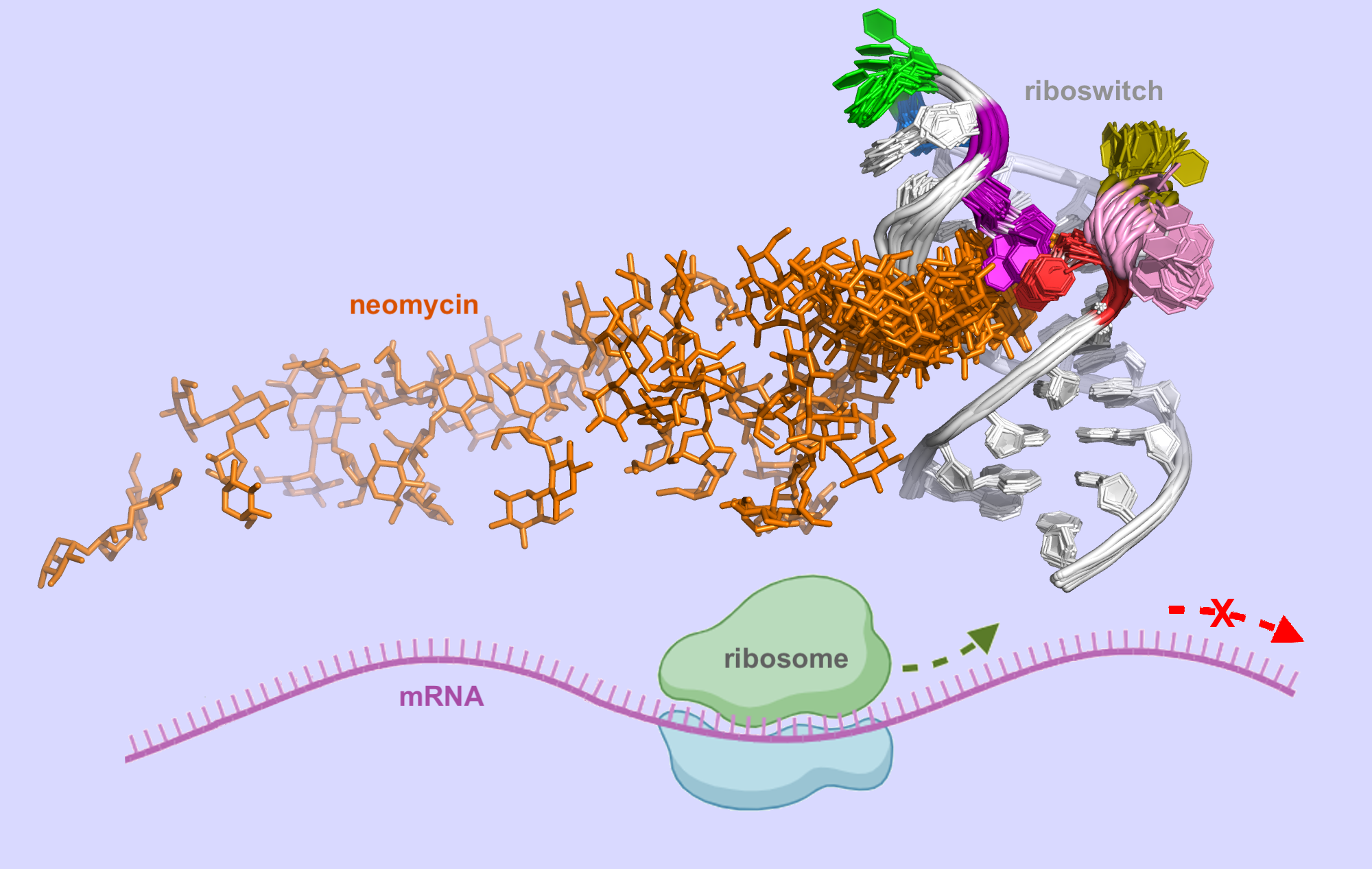
Naukowcy zbadali syntetyczny ryboprzełącznik, który wiąże cząsteczkę neomycyny. Opracowali metodę i narzędzia do opisu ścieżki wiązania neomycyny z tym segmentem mRNA. Wyniki ich symulacji pozwoliły zrozumieć mechanizm działania tego ryboprzełącznika. Może to pomóc w projektowaniu syntetycznych ryboprzełączników i cząsteczek, które kontrolują ich dynamikę i strukturę.
– Nasza praca pokazuje, jak kluczowe etapy oddziaływania neomycyny z ryboprzełącznikiem mogą być zoptymalizowane, aby zwiększyć jego skuteczność regulacyjną w komórkach. Wyniki symulacji pokazują istotne różnice w ścieżce wiązania neomycyny do różnych sekwencji ryboprzełącznika oraz wskazują, jak te różnice wpływają na jego aktywność – dodaje prof. Trylska.
Przeciw komórkom bakteryjnym
Opracowana przez badaczy metodologia i narzędzia mogą wspomóc proces projektowania nowych leków poprzez wybór najbardziej obiecujących związków do dalszych badań doświadczalnych. Wyniki badań naukowców z UW oraz instytutu RIKEN mogą przyczynić się do przyspieszenia projektowania fragmentów RNA jako potencjalnej strategii w walce z opornością mikroorganizmów na antybiotyki.
Piotr Chyży, Marta Kulik, Ai Shinobu, Suyong Re, Yuji Sugita, Joanna Trylska*, Molecular dynamics in multidimensional space explains how mutations affect the association path of neomycin to a riboswitch, „Proceedings of the National Academy of Sciences”. Tekst artykułu znajduje się na stronie PNAS.